 打印
打印
在国家自然科学基金项目(批准号:62473212、62203236)等资助下,南开大学陈盛泉教授团队在面向细胞类型识别的多源组学数据智能整合方面取得进展,研究成果以“Rigorous integration of single-cell ATAC-seq data using regularized barycentric mapping”为题,于2025年8月26日发表于《自然•机器智能》(Nature Machine Intelligence)上。论文链接:https://www.nature.com/articles/s42256-025-01099-3。
随着高通量测序技术的发展,如何从海量、高维、多源的单细胞组学数据中提取有价值的生物学信息,并实现准确的细胞类型识别,已成为计算生物学与数据科学领域的一项关键挑战。单细胞染色质开放性测序(scATAC-seq)数据具有高维度(百万级特征)、高稀疏度(约1%非零)、强噪声及分布二值化等复杂特性。此外,由于多数据源引入的技术噪声(即批次效应)常与生物异质性高度耦合,导致当前的数据整合方法往往难以区分二者,易出现“过度校正”问题,即在消除批次偏差的同时,也抹除了真实的生物学差异。
针对上述问题,研究团队提出了基于正则化重心映射的深度学习框架Fountain。该框架首先通过深度神经网络将高维、稀疏的原始数据投影到一个能捕捉其核心特征的紧凑低维隐空间。在隐空间中,Fountain将不同批次数据“迁移”到一个统一的参考批次中,实现多源数据的智能整合。在迁移过程中,为防止细胞间原有的生物学关系被“扭曲”破坏,Fountain引入了一个几何正则化项,以确保细胞在各自批次内部的局部几何结构(即细胞间的相似性关系)在整合后得以保留,从而有效区分技术噪声与真实生物信号,避免过度校正。
大量基准测试结果表明,Fountain在细胞类型识别准确性及批次效应校正效果方面均优于当前主流方法。在计算效率上,Fountain展现出优异的可扩展性,能够高效处理百万级细胞的大规模数据集。该方法还支持在线批次整合:模型经首次训练后,可直接用于新生成批次数据的整合,为构建可动态扩展与更新的细胞图谱提供了关键技术支持。此外,Fountain还能够输出经过校正与增强、且与原始数据维度一致的矩阵,为下游更精细的生物学分析提供了高质量的数据基础。
该研究不仅为单细胞组学提供了先进的数据整合与细胞识别工具,其在高维稀疏数据处理、最优传输与流形学习的融合以及模型泛化能力提升等方面的算法思路,也为信息科学中其他复杂数据分析任务提供了新借鉴。
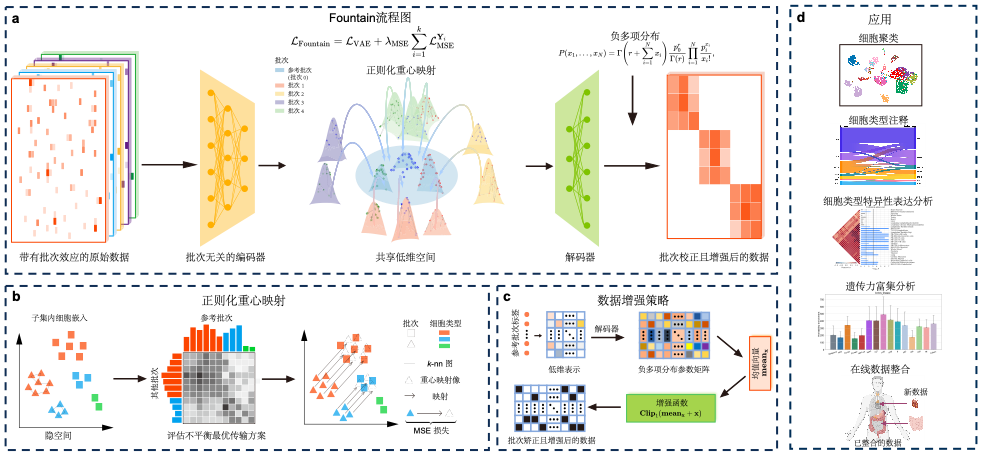
图 Fountain方法有效整合多源组学数据并准确识别细胞类型
