资助成果
我国学者在细胞膜表面分子发现与适体组学研究方面取得进展
日期:2026-01-09
来源: 化学科学部
作者:蔡彬 王勇
字号:【
大
中
小】
 打印
打印
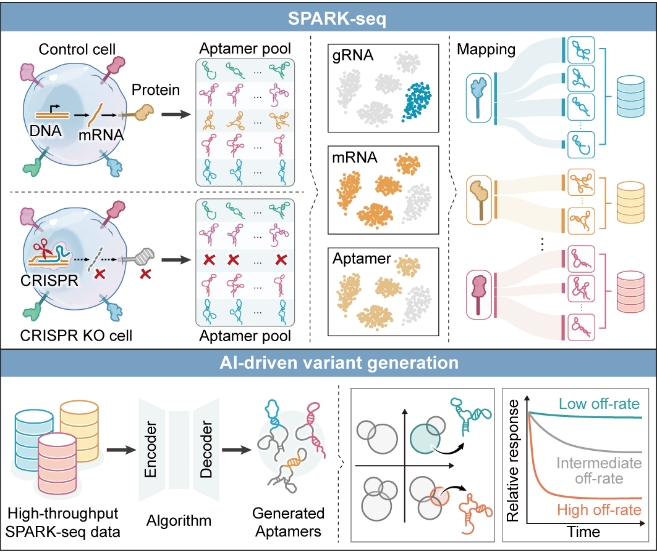
图 SPARK-seq用于核酸适体发现与序列变体设计。(上)Spark-seq工作流程,(下)基于人工智能的核酸适体理性设计
在国家自然科学基金项目(批准号:22474075、22304177、T2188102)等资助下,中国科学院杭州医学研究所谭蔚泓与吴芩团队在细胞膜表面分子发现的研究方面取得进展,研究成果以“SPARK-seq: 高通量适体发现与动力学表征平台(SPARK-seq: A high-throughput platform for aptamer discovery and kinetic profiling)”为题,于2026年1月1日发表于《科学》(Science)杂志。论文链接https://doi.org/10.1126/science.adv6127。同期《科学》发表评论文章,以“点燃核酸适体数据科学的火花(Light the spark of aptamer data science)”为题,评价该研究在数据驱动的核酸适体科学中所起的引领作用。
细胞表面约60%的药物靶点是膜蛋白,却因膜蛋白质组复杂、候选量巨大而使癌症标志物筛选难度陡增,验证周期漫长且效率低。针对该挑战,上述研究团队开发出一种高通量适体发现与动力学表征平台,简称SPARK-seq。该平台融合了CRISPR基因编辑、核酸适体组学与单细胞多组学技术,在单细胞层面同步实现“靶点扰动‑表型读取‑探针结合”的全流程解析。利用该技术,该团队在单次实验中系统分析了8000余个单细胞,从5000多条序列中发现8种肿瘤关键膜蛋白的特异性探针,并覆盖了丰度差异百倍以上的靶点。该方法在天然细胞环境中进行原位筛选,避免了传统体外筛选中的构象失真问题,提升了探针的临床转化潜力。

下一篇:
我国学者在相变储热研究领域取得进展