 打印
打印
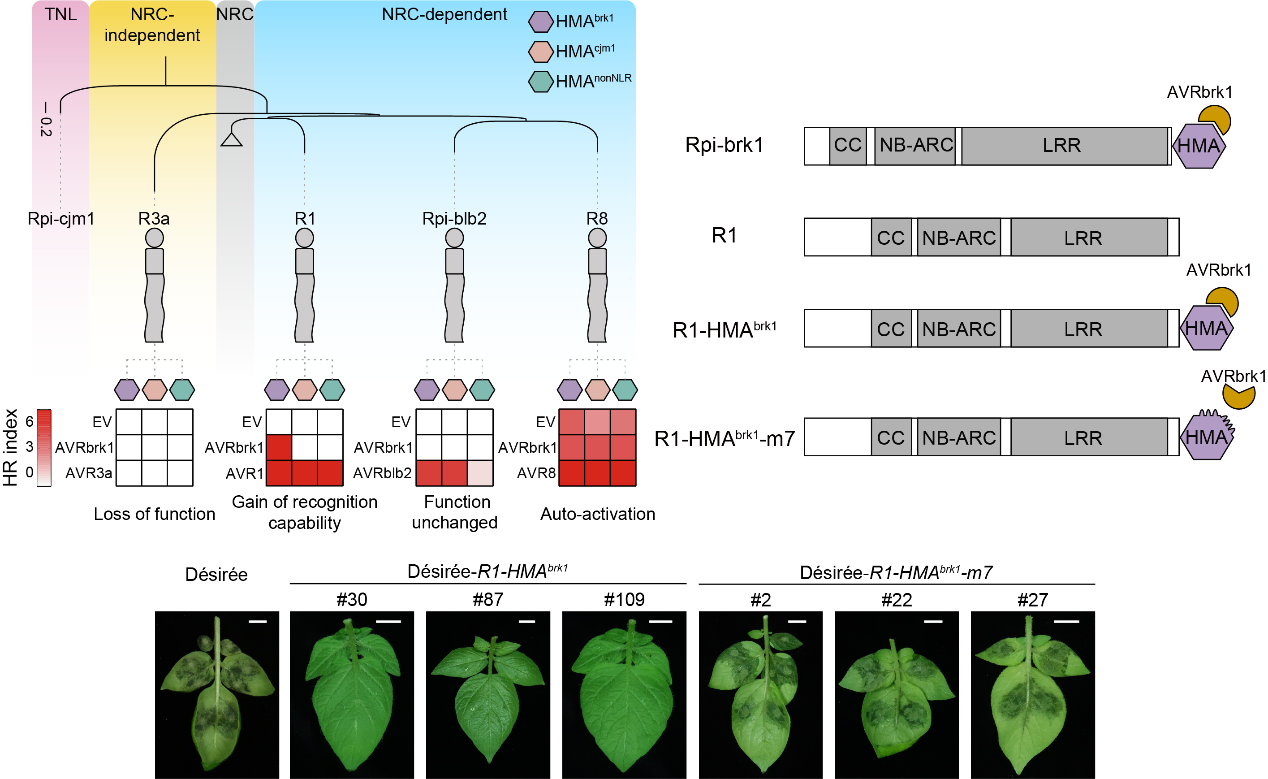
图 “Plug-in“抗病育种策略提高马铃薯晚疫病抗性
在国家自然科学基金卓越研究群体项目(批准号:32488302)等资助下,中国农业科学院深圳农业基因组研究所黄三文团队和南京农业大学董莎萌团队在马铃薯抗病育种领域取得新进展,构建了马铃薯抗病基因资源库,揭示了识别和辅助两类抗病基因在序列和功能上的分化特征,并阐明了它们与具有不同进化潜力的病原菌之间的协同演化规律。利用比较和演化基因组学,研究团队挖掘出三个新抗晚疫病基因,并在对其抗病机理的深入解析基础上提出了“Plug-in(插件式)”抗病育种新策略,为培育持久抗病的马铃薯品种开辟了全新路径。研究成果以“Plug-in Strategy for Resistance Engineering Inspired by Potato NLRome(马铃薯抗病基因组助力插件式抗病设计育种)”为题,于2025年10月30日在线发表于《自然》(Nature)。论文链接:https://www.nature.com/articles/s41586-025-09678-5。
马铃薯晚疫病是全球重大农作物病害之一,也是被农业农村部列入我国《一类农作物病虫害名录》的8个重大病害之一,其爆发曾造成史上著名的“爱尔兰大饥荒”,每年造成近百亿美元损失。由于病原菌致病疫霉的基因组结构复杂,变异迅速,目前农业育种中使用的抗病基因快速失去作用,亟待进一步挖掘新的晚疫病抗性基因资源。
传统马铃薯育种依赖四倍体品种,遗传背景复杂,抗病基因难以有效聚合。为解决上述难题,黄三文团队联合国内外优势团队发起了“优薯计划”,提出以二倍体替代四倍体、以杂交种子替代薯块的策略,从根本上变革马铃薯的育种和繁殖方式,而晚疫病抗病育种是“优薯计划”的重要研究方向。
研究通过精细注释马铃薯中的抗病基因,构建了包含近4万个抗病基因的“马铃薯抗病基因组(NLRome)”,建立了目前植物界最完整、规模最大的抗病基因资源库,系统分析并揭示了抗病基因进化与功能的关系,为抗病基因功能预测提供了演化依据。依托上述基因组资源,团队利用比较和演化基因组学方法,成功克隆三个新型马铃薯抗晚疫病基因。研究还发现将抗病基因Rpi-brk1的功能结构域HMA“嫁接”到已失效的抗晚疫病蛋白R1的末端后,可使其重新获得抗性功能。该研究系统研究了马铃薯中抗病基因的进化规律,提出抗病基因快速克隆的可行案例,首次提出“即插即用”的抗性基因设计路线为快速培育广谱抗病品种提供了全新思路。
