——我国学者提出基于语言模型的蛋白趋同演化分析方法
 打印
打印
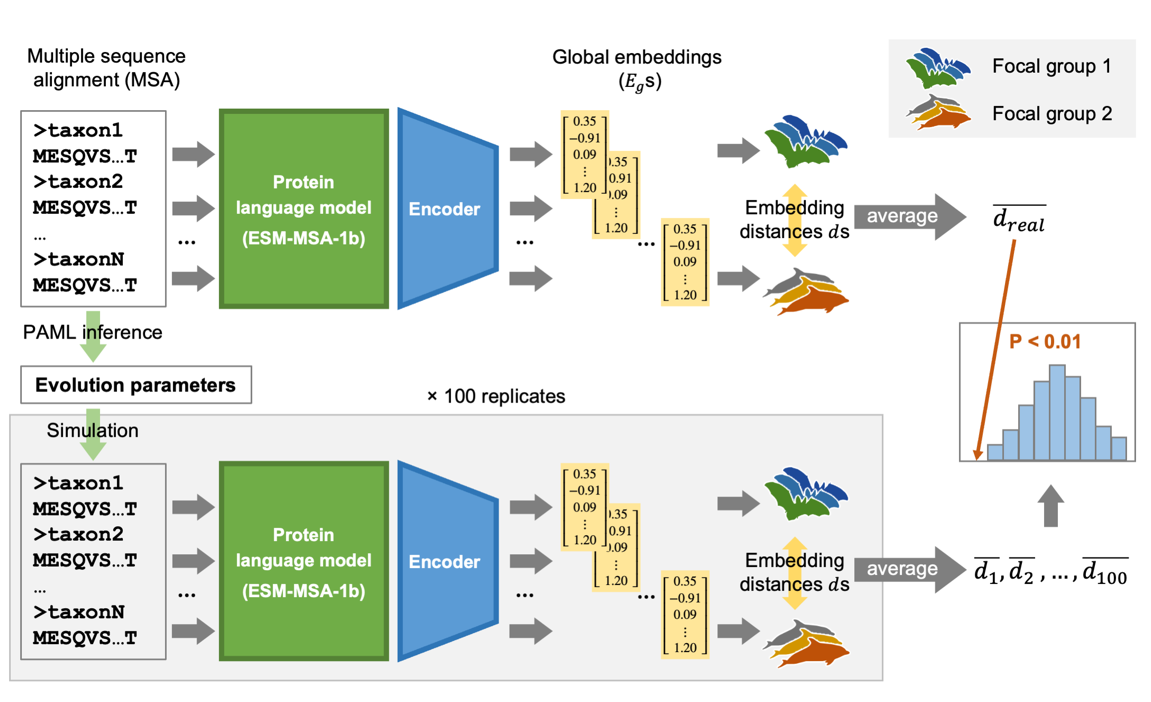
图 ACEP流程示意图以及在回声定位哺乳类中检测出的候选基因
在国家自然科学基金项目(批准号:92370134)等资助下,中国科学院动物研究所邹征廷研究员团队提出基于语言模型的分子序列演化分析新范式,利用预训练蛋白语言模型,揭示了蛋白高阶特征在功能适应性趋同演化中的重要作用。研究成果以“Language models reveal a complex sequence basis for adaptive convergent evolution of protein functions”为题,于2025年9月23日在线发表在国际学术期刊《美国国家科学院院刊》(PNAS)上,论文链接:https://doi.org/10.1073/pnas.2418254122。
趋同演化指不同类群的生物在适应相似环境的过程中,独立演化出相似的功能性状,是生物演化中的重要现象。长期以来,演化生物学领域致力于探索表型趋同背后的分子机制。传统研究方法多聚焦于蛋白序列中单个氨基酸位点的趋同变化;然而,越来越多的证据表明,即使没有明确的位点趋同,同源蛋白仍可能通过高阶结构或理化特征的趋同演化实现功能上的相似性。
为此,研究团队提出使用蛋白语言模型来捕捉序列中复杂的上下文信息和高阶特征,并设计了ACEP分析流程,利用预训练蛋白语言模型计算同源蛋白嵌入向量的真实距离,并模拟中性演化过程构建背景距离分布,从而进行统计检验。ACEP在回声定位哺乳动物等多个经典案例中展现出显著结果,识别出了已知的和新的适应性趋同演化候选基因,如CIB2、GSN等,并展现出可解释的具体理化特征趋同模式。该研究不仅揭示了蛋白高阶特征趋同是适应性演化的重要机制,也展示了人工智能技术在针对复杂的基因型-表型映射进行演化生物学分析时的潜力。
