|
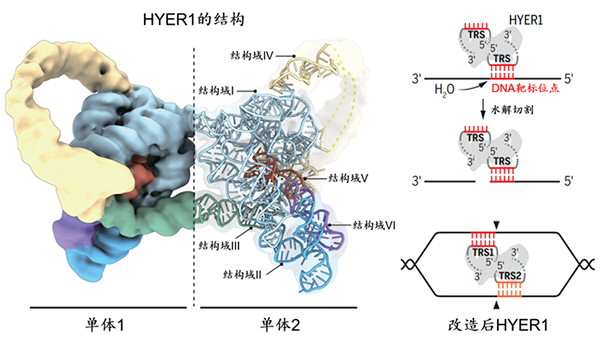 |
图 具有DNA靶向切割能力的HYER1的结构和DNA水解切割示意图
在国家自然科学基金项目(批准号:32150018)等资助下,清华大学刘俊杰课题组在基于RNA核酶的基因编辑工具设计和开发方面取得新进展。研究成果以“可编程的水解型核酸内切核酶用于DNA靶向切割(Hydrolytic endonucleolytic ribozyme (HYER) is programmable for sequence-specific DNA cleavage)”为题,于2024年2月2日在线发表在《科学》(Science)杂志上,论文链接:https://www.science.org/doi/10.1126/science.adh4859。
基因承载着生命的遗传密码,定义了生命的运行法则。基因的靶向编辑,在基因功能研究,生物性状操纵,生物工程化改造等场景中,扮演着不可或缺的重要角色。目前常用的基因编辑工具均以蛋白核酸酶为基础,如ZFNs、TALENs和CRISPR-Cas系统等,具有组分复杂、分子体积较大、组织导入效率低以及存在免疫原性等缺陷。该团队关注于第二类内含子,一种具有转座能力的RNA-蛋白复合物。第二类内含子可以通过RNA引导识别DNA底物,并通过蛋白对DNA进行切割以及完成逆转录,从而实现在宿主基因组中的扩增。然而,该团队发现了大量不含蛋白组分的第二类内含子也在宿主基因组中存在多拷贝的现象,暗示着其潜在的DNA切割活性。这为挖掘具有DNA切割活性的RNA核酶分子提供了可能的方向,进而有望开发出基于RNA,且具有我国核心自主知识产权的全新基因编辑平台。
该团队结合了多学科交叉领域技术,如基于协方差模型的高通量RNA核酶分子筛选技术、基于冷冻电子显微镜的生物大分子高分辨结构解析技术,以及RNA核酶分子的体外和体内活性验证技术等。利用这些技术,他们构建了一套完善的实验流程,能够精准、高效地大规模挖掘RNA核酶分子,并在体外实验系统和细胞内进行DNA切割活性验证。进一步,他们针对高活性的RNA核酶分子,进行结构解析,并依据所得高分辨三维结构,成功完成了多种理性设计,提高了RNA核酶分子的效率和特异性。
该研究系统性地发现了多个具有DNA靶向水解切割活性的RNA核酶分子(HYERs),并在体外系统、大肠杆菌和哺乳动物细胞中证明了其DNA切割能力。此外,研究还揭示了高活性的HYER1分子以同源二聚体的形式存在,并阐明了DNA水解切割过程的分子机制。基于高分辨结构信息,该团队进一步对RNA核酶进行了延长底物识别序列、引入新的底物识别基元、构建异源二聚体等多种理性设计,成功提高了DNA底物识别特异性,拓展了双链DNA底物的切割模式。这项研究不仅极大地扩充了RNA核酶分子数据库,更新了科学界对RNA功能的认知,也为开发基于RNA的全新基因编辑平台提供了坚实基础,有望引领基因编辑技术从“以蛋白质为基础”转变为“以RNA为基础”的时代,并逐步扩展到其它分子生物技术领域,具有重要的研究意义和价值。