|
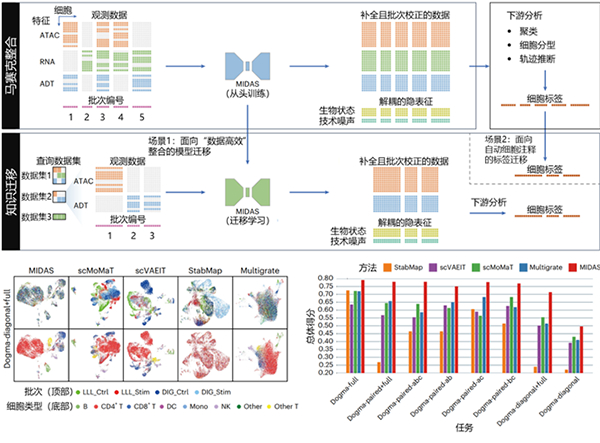 |
图 MIDAS的功能概览与性能比较
在国家自然科学基金项目(批准号:62303488)等资助下,军事科学院军事医学研究院应晓敏教授团队和伯晓晨教授团队提出了一种用于单细胞多组学数据马赛克整合及知识迁移的计算工具MIDAS。研究成果以“利用MIDAS进行单细胞多模态数据的马赛克整合和知识迁移(Mosaic integration and knowledge transfer of single-cell multimodal data with MIDAS)”为题,于2024年1月23日发表于《自然•生物技术》(Nature Biotechnology)期刊上。论文链接:https://www.nature.com/articles/s41587-023-02040-y。
单细胞测序技术是近年来生命科学领域的突破性技术,能够检测单个细胞内的多种遗传物质与功能分子(例如RNA表达、蛋白质丰度和染色质可及性),从而揭示细胞在不同分子层面的异质性以及组学之间的关联,有助于更加深入地理解细胞功能,探索生命发育和疾病发生等机制。然而,随着测序技术的发展和测序数据的增长,不同组学组合、不同测序技术、不同测序样本的“马赛克”式单细胞数据的整合成为巨大的挑战。
为此,应晓敏团队和伯晓晨团队提出了基于生成式人工智能的新方法MIDAS。MIDAS假设每个细胞的多模态观测值是通过深度神经网络从两个与模态无关且解耦的隐变量生成的(即代表细胞异质性的生物状态,以及由单细胞实验引起的技术噪声),其输入由不同单细胞样本(批次)的表达矩阵和批次编号向量组成。这些批次可能来自不同实验或不同测序技术(例如CITE-seq和ASAP-seq),因此可能存在不同的技术噪声、模态组合和观测特征。MIDAS的输出包括生物状态和技术噪声两种低维表示的矩阵,以及对缺失模态和特征进行了补全并消除了批次效应的表达矩阵。这些输出可以用于聚类、细胞分型、轨迹推断等下游分析。
UMAP可视化结果和定量评估结果表明MIDAS消除了批次效应,很好地保留了生物信号,在不同的马赛克任务上表现稳定,性能显著优于国际同类算法,如图。此外,MIDAS可以高效而灵活地将参考数据集中的知识迁移到查询数据集中,从而方便处理新的单细胞多组学数据,且基于MIDAS降维后的隐变量也可以对模态缺失的马赛克数据进行拟时序分析,当在不同组织之间迁移知识时,MIDAS在对齐异构数据集、识别已知细胞类型、发现未知细胞类型等方面表现出优秀的性能。
综上,MIDAS对于揭示细胞的功能和分子调控机制、研究疾病的发生发展过程具有重要意义,预期为疾病诊断与精准治疗等应用提供有力的技术支撑。