|
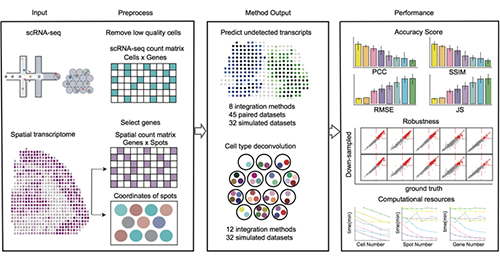 |
图 空间转录组和单细胞转录组数据整合算法性能比较分析流程
在国家自然科学基金项目(批准号:T2125012)的资助下,中国科学技术大学瞿昆教授课题组通过设计一整套分析流程,系统性评估了16种空间转录组和单细胞转录组数据整合算法在预测基因或细胞类型空间分布方面的性能。相关成果以“用于转录本分布预测和细胞类型拆分的空间转录组和单细胞转录组整合分析算法性能比较(Benchmarking spatial and single-cell transcriptomics integration methods for transcript distribution prediction and cell type deconvolution)”为题发表在《自然·方法》(Nature Methods)杂志上。论文链接为:https://doi.org/10.1038/s41592-022-01480-9。
细胞在组织器官内所处的空间位置对于它发挥特异性功能至关重要。近年来,研究者开发了多种空间转录组技术可以在保留细胞精确空间定位的同时,检测细胞内全转录组的表达,以此来研究在发育或疾病发生发展过程中起关键性作用的细胞亚群及其分子机制。
现有的空间转录组学技术存在两个不足:1. 基于测序的空间转录组技术无法实现真正意义上的单细胞分辨率;2. 基于成像空间转录组的技术能检测到的基因通量有限。为了突破技术的局限性,生物信息学家设计了多种算法整合空间转录组与单细胞转录组数据,预测细胞类型的空间分布和/或单个细胞的完整转录组信息。这些算法大大加深了我们对空间转录组学数据及相关生物学和病理过程的理解。然而,由于不同算法的工作原理和适用范围存在显著差异,使得研究者很难选择最佳算法用来预测细胞类型和基因表达的空间分布。
瞿昆教授课题组长期致力于开发生物大数据分析算法和软件。在此项研究中,课题组收集了45对同一组织来源的空间转录组与单细胞转录组数据集,和32个模拟数据集,并设计了多种指标,从准确性、鲁棒性、计算资源耗时等多维度系统性评估了16种整合算法的性能。研究结果显示,Cell2location、SpatialDWLS和RCTD算法能更加准确的预测细胞类型的空间分布;Tangram、gimVI和SpaGE算法是预测基因表达空间分布的最佳算法。Tangram、Seurat和LIGER算法计算效率相对较高,适合处理大规模数据集(图)。
该研究工作总结了每种算法的属性、性能、适用性以及高效算法的优势,为研究人员进一步提升算法性能提供了参考。此外,该研究在github上提供了整合空间转录组和单细胞转录组数据的分析流程,为研究人员提供了数据处理的分析工具。