国家蛋白质科学中心徐平研究员、贺福初院士团队合作揭示不同泛素链类型的转换介导转录因子的激活
近日,来自军事医学研究院生命组学研究所和国家蛋白质科学中心的徐平研究员、贺福初院士和来自美国加利福尼亚大学尔湾分校的Peter Kasier教授团队合作,在著名学术期刊Molecular Cell上发表文章“Proteomics Links Ubiquitin Chain Topology Change to Transcription Factor Activation”,该研究发现K11泛素链参与了甲硫氨酸转录因子Met4的转录激活调控。1

作为真核细胞内普遍存在的一种翻译后修饰,泛素化修饰参与了真核生物生长、发育、分化及衰老等几乎所有的生理过程的调控,在内环境稳态维持中发挥了重要的调控作用。泛素化及其信号途径的任何突变或紊乱都可能诱发如癌症、神经退行性疾病等多种严重疾病。因此,深入研究泛素化信号的调控机制,对于认识生命活动、理解致病分子机制并研发相应治疗方法具有重要意义。
泛素分子的7个赖氨酸残基和N末端甲硫氨酸均可作为泛素继续链接的位点,形成8种拓扑形式且长度各异的同质泛素链。当底物蛋白上同一赖氨酸位点的泛素链修饰是由多种不同的泛素链混合延伸而成时就会生成混合或者分支泛素链修饰,即异质泛素链。这些同质或异质泛素链拓扑结构各异;随着泛素链长度的延伸,其拓扑结构的复杂性呈现指数型增长。各异拓朴结构的泛素链赋予了底物蛋白以新的功能,介导了同一分子上不同信号之间的转换,形成了严密调控的“泛素密码”2。
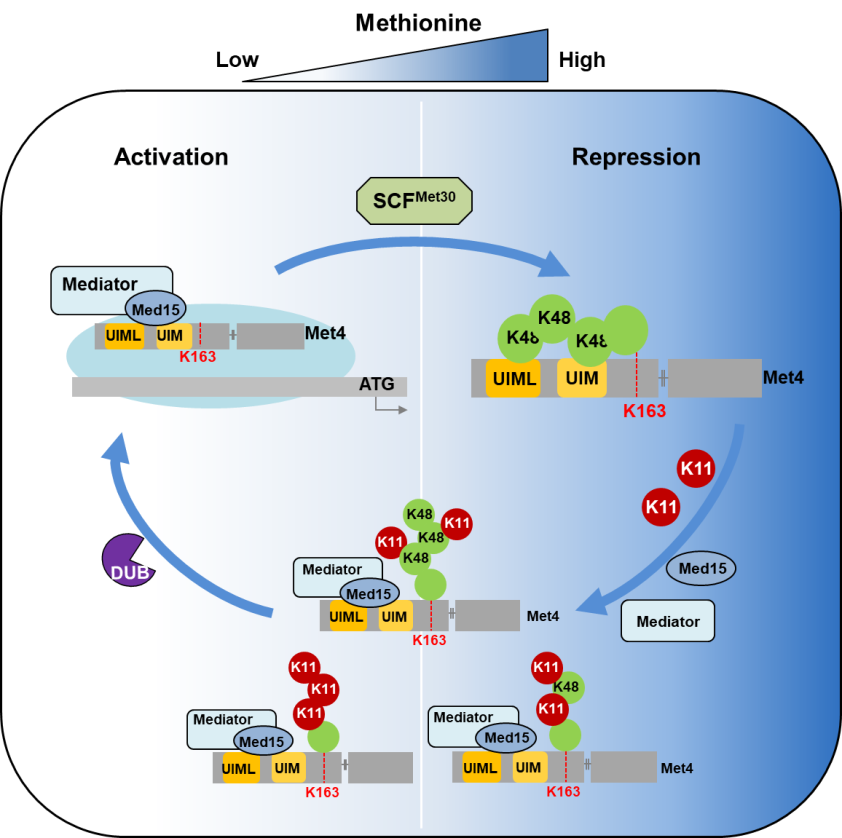
以研究最清楚的经典泛素链K48泛素链为例,常规条件下,它主要介导了底物蛋白的蛋白酶体降解过程。然而,随着研究的拓展与深入,科学家发现K48泛素链还具有非蛋白降解的功能。典型的例子是K48链作为转录抑制因子参与了酵母细胞甲硫氨酸合成通路相关转录因子Met4的转录激活调控、甲硫氨酸代谢和细胞增殖等过程3-4。然而,当细胞处于甲硫氨酸匮乏的环境中,Met4如何由失活状态转变为活化状态的分子机制仍处于空白状态。
本研究通过高覆盖定量蛋白质组学技术实现了对酵母蛋白组的深度覆盖,在此基础上通过SILAC代谢标记技术比较了野生型和泛素K11位点突变为R的突变型菌株(K11R)的蛋白组表达差异,发现K11泛素链的缺失可引起甲硫氨酸合成通路所有合成酶蛋白水平的显著下调。考虑K11泛素链的缺失并不影响该通路关键转录因子Met4本身的蛋白稳定性,研究者证明了K11泛素链参与对Met4转录激活状态的调控。此研究揭示了K11泛素链一种全新的生理功能,即作为一种信号开关,通过底物蛋白上修饰泛素链种类以拓朴结构的转换,实现生物学信号的改变及转录因子活性的转变,完成相关基因转录激活。该项研究深入探究了甲硫氨酸合成通路关键转录因子Met4由失活到激活状态转变的分子机制,也为泛素链作为分子开关转换信号传递方向提供了新的例证,为复杂且精密的泛素密码的解析提供了理论依据。此外,该项研究也为目前有限的泛素化修饰对转录因子活性状态的调控机制探究提供了理论基础。
国家蛋白质科学中心李衍常博士、高媛博士、武汉大学和国家蛋白质科学中心联合培养的博士生兰秋艳、国家蛋白质科学中心的博士生张成普、美国Emory大学神经推行性疾病中心的Eric B. Dammer博士和Duc Duong以及美国加州大学尔湾分校的Mark A. Villamil是论文的第一作者。该研究得到了国家自然科学基金以及科技部重点研发计划等基金的资助。
论文链接:https://www.sciencedirect.com/science/article/abs/pii/S1097276519305040
参考文献:
1.Li, Y.; Dammer, E. B.; Gao, Y.; Lan, Q.; Villamil, M. A.; Duong, D. M.; Zhang, C.; Ping, L.; Lauinger, L.; Flick, K.; Xu, Z.; Wei, W.; Xing, X.; Chang, L.; Jin, J.; Hong, X.; Zhu, Y.; Wu, J.; Deng, Z.; He, F.; Kaiser, P.; Xu, P., Proteomics Links Ubiquitin Chain Topology Change to Transcription Factor Activation. Molecular Cell 2019.
2.Komander, D.; Rape, M., The ubiquitin code. Annu Rev Biochem 2012, 81, 203-29.
3.Su, N. Y.; Ouni, I.; Papagiannis, C. V.; Kaiser, P., A dominant suppressor mutation of the met30 cell cycle defect suggests regulation of the Saccharomyces cerevisiae Met4-Cbf1 transcription complex by Met32. J Biol Chem 2008, 283 (17), 11615-24.
4.Kaiser, P.; Flick, K.; Wittenberg, C.; Reed, S. I., Regulation of transcription by ubiquitination without proteolysis: Cdc34/SCFMet30-mediated inactivation of the transcription factor Met4. Cell 2000, 102 (3), 303-314.
